Nauka
Bakterie i ich wirusy: wyścig zbrojeń przerwany tysiące lat temu
Dobrze zachowane DNA kilkuset gatunków bakteriofagów sprzed ponad tysiąca lat – wirusów infekujących bakterie – znaleziono w próbkach ludzkich odchodów i wnętrznościach starożytnej mumii. Okazuje się, że genom jednego z gatunków jest prawie taki sam jak genom współczesnych wirusów. Dlaczego ewolucja tego gatunku zatrzymała się na tak długo?
Wyniki badań międzynarodowego zespołu pod przewodnictwem polskich naukowców, opublikowane w Komunikacja przyrodniczazmienić postrzeganie „wyścigu zbrojeń” pomiędzy wirusami a ich bakteryjnymi gospodarzami.
„To, co odkryliśmy, wydaje się zaprzeczać naszej wcześniejszej wiedzy na temat bakteriofagów” – powiedział PAP pierwszy autor badania, Piotr Rozwalak z Uniwersytetu im. Adama Mickiewicza w Poznaniu. Badania zostały przeprowadzone w ramach jego pracy magisterskiej.
Aby się rozmnażać, wirusy muszą przedostać się do komórki gospodarza i zmusić ją do skopiowania materiału genetycznego. Istnieją wirusy atakujące komórki roślinne, ludzkie i zwierzęce. Infekcje wirusowe zagrażają także komórkom bakteryjnym. Te wirusy specyficzne dla bakterii to bakteriofagi (w skrócie fagi).
Zarówno bakterie, jak i bakteriofagi zamieszkują ludzki układ trawienny. Niektóre są korzystne lub neutralne dla zdrowia ludzkiego, inne niekoniecznie. Jego DNA można znaleźć w próbkach kału lub w materiale pobranym z jelita.
Ekspert bioinformatyki i współautor pracy dr Andrzej Zieleziński z UAM stwierdza, że dzięki osiągnięciom metagenomiki możliwe jest sekwencjonowanie i odczytywanie fragmentów DNA znajdujących się w próbce materiału biologicznego pobranego z dowolnego środowiska ( np. gleba lub układ trawienny człowieka). Te wycięte „fragmenty układanki” można złożyć w większe fragmenty, w tym całe genomy, i porównać ze znanymi już genomami różnych organizmów.
Umożliwia to sekwencjonowanie milionów fragmentów łańcuchów DNA zawartych w próbce materiału, nawet jeśli próbka ma tysiące lub miliony lat, ale jest doskonale zachowana.
RECYKLING PALEOFAJTU
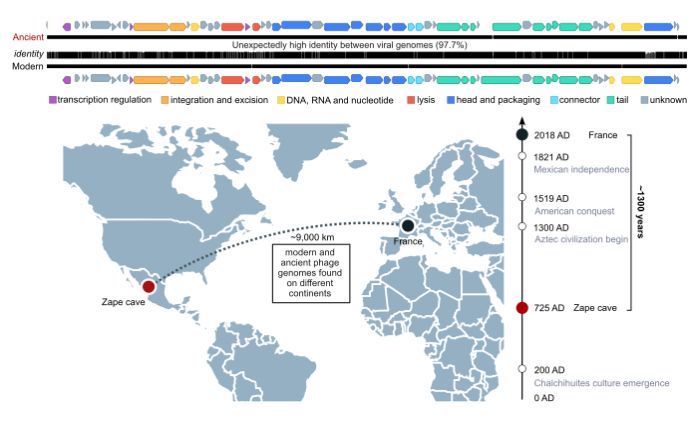
W ten sposób zespoły z całego świata zbadały ludzkie odchody sprzed ponad tysiąca lat, zachowane w pustynnych jaskiniach Meksyku i Stanów Zjednoczonych (Arizona i Utah). Pobrano także próbki z jelit 5000-letniej mumii ludzkiej znalezionej w Alpach lub w kopalni soli z epoki brązu.
Co ciekawe, dane z tych badań były publicznie dostępne. Aby uzyskać dostęp do danych na temat kopalnego DNA zawartego w paleofach, nie było potrzeby przeprowadzania kosztownych badań, badań archeologicznych i sekwencjonowania genomu; wystarczyło przeanalizować ogólnodostępne bazy danych i poszukać informacji innych niż początkowo poszukiwali ich autorzy.
„Przeprowadziliśmy ten recykling danych” – mówi Piotr Rozwalak.
Podczas gdy poprzednie badania innych zespołów skupiały się na wielowiekowych bakteriach występujących w florze jelitowej, zespół kierowany przez polskich naukowców zdecydował się skupić na genomie pradawnych bakteriofagów zamieszkujących układ pokarmowy człowieka. «W ten sposób zrobiliśmy krok w kierunku nowej dziedziny wiedzy, wirusomiki, czyli badania wirusów obecnych w różnych próbkach środowiskowych. Jako jedni z pierwszych zbadaliśmy próbki na obecność bakteriofagów. Otwieramy drzwi do zupełnie nowej dziedziny, jaką jest wirusomika kopalna, czyli badanie wirusów z przeszłości” – mówi Rozwalak.
WYŚCIG ZBROJEŃ
Naukowcy postanowili sprawdzić, jak szybko zmieniał się genom wirusów bakteryjnych na przestrzeni ostatnich kilku stuleci. Założyli, że przynajmniej niektóre bakteriofagi kopalne będą podobne do współczesnych. Zdali sobie sprawę, że przeszkodą może być proces ewolucyjny, który zatrze wszelkie ślady podobieństwa pomiędzy kolejnymi generacjami wirusów. To nieustanny wyścig zbrojeń pomiędzy bakteriami i wirusami, który szybko może zmienić obie strony nie do poznania.
Bakteriofagi często przyczyniają się do śmierci lub osłabienia bakterii. Dlatego ogólnie w interesie bakterii leży wytworzenie skutecznych mechanizmów obronnych przed ich patogenami. Jeśli bakteria znajdzie sposób na obronę przed infekcją, może przekazać tę genetyczną „aktualizację oprogramowania” swoim sąsiadom. A wtedy będą się rozmnażać tylko wirusy, które znajdą nową lukę w obronie gospodarza. Dlatego wydawało się, że bakteriofagi i ich gospodarze zawsze ewoluowały bardzo szybko.
I tu pojawiła się niespodzianka. Naukowcy odkryli genomy prawie 300 gatunków bakteriofagów, które były częściowo podobne do tych spotykanych obecnie. Jeden z wirusów był jednak wyjątkowy: jego sekwencja genomowa była w 97,7% podobna do genomu współczesnego bakteriofaga. Ten wirus nazywa się wirus mushu. Zaraża beztlenowe bakterie jelitowe, które korzystnie wpływają na nasze zdrowie m.in Faecalibacterium płeć.
NIEZMIENNIE
Co sprawiło, że potomkowie wirusa wyglądają tak samo jak ich przodkowie sprzed 1300 lat? Wskazuje to na „długoterminowy związek profaga z jego żywicielem” – piszą autorzy publikacji.
Rozwalak mówi: „Nasz wirus jest wbudowany w genom bakterii. I z jakiegoś powodu bakteria nie może się przed tym obronić. Nasza hipoteza jest taka, że istnieje relacja mutualistyczna: każda ze stron w jakiś sposób czerpie korzyści z obecności drugiej strony – coś otrzymuje i chce współpracować. Dlatego na podstawie tych badań można założyć, że czasami bakterie mogą współpracować z fagami i czerpać korzyści z ich obecności.
Dr Zieleziński sugeruje dodatkowo, że stabilność wirusa może wynikać także z jego zdolności do infekowania aż ośmiu różnych gatunków bakterii. A to dowodzi, że wirusy są bardzo wszechstronne. Jednak w takiej sytuacji istotne zmiany w wirusowym DNA mogą prowadzić do utraty zdolności do zakażania jednego z możliwych gospodarzy. Zatem wirus nie jest zainteresowany ewolucją.
OPŁATY BADAWCZE
Zieleziński wyjaśnia, że przy badaniu kopalnego DNA należy zachować ogromny sceptycyzm, gdyż istnieje możliwość przypadkowego skażenia próbek współczesnym DNA. Dlatego autorzy przeprowadzili serię rygorystycznych testów, aby zweryfikować autentyczność wirusa kopalnego. Wyniki testów wyraźnie to pokazały wirus mushubył tak stary, jak badana próbka.
„Przeprowadziliśmy pierwszą na świecie rekonstrukcję genomów kopalnych bakteriofagów w próbkach reprezentujących środowisko jelitowe człowieka sprzed nawet 5000 lat i stworzyliśmy katalog tych wirusów, rzucając nieco światła na nieznane wcześniej aspekty historii mikroświata jelitowego. W ten sposób odkryliśmy gatunek wirusa, który nie tylko przetrwał wieki w ludzkim jelicie, ale także zachował swoją formę w niezmienionej formie przez co najmniej 1300 lat” – mówi dr.
PAP – Nauka w Polsce, Ludwika Tomala
lt/ agt/ kap/
tr. RL
Fundacja PAP umożliwia nieodpłatne przedrukowanie artykułów z portalu Nauka w Polsce pod warunkiem otrzymania raz w miesiącu wiadomości e-mail o korzystaniu z portalu i wskazaniu pochodzenia artykułu. Na stronach internetowych i portalach internetowych należy podawać adres: Źródło: www.scienceinpoland.pl, natomiast w czasopismach – adnotację: Źródło: Nauka w Polsce – www.scienceinpoland.pl. W przypadku serwisów społecznościowych należy podać jedynie tytuł i lead przesyłki naszej agencji wraz z linkiem prowadzącym do tekstu artykułu na naszej stronie, tak jak widnieje on na naszym profilu na Facebooku.

„Piwny maniak. Odkrywca. Nieuleczalny rozwiązywacz problemów. Podróżujący ninja. Pionier zombie. Amatorski twórca. Oddany orędownik mediów społecznościowych.”
